概要
超遠心分析とは、タンパク質溶液を高速で遠心し、遠心力場におかれたタンパク質分子が溶媒の中を沈降する様子をリアルタイムで観測・記録することによって、タンパク質の均一性を検定すると共に分子量を求める手法である (1)。この手法の特徴として、通常の水溶液中のタンパク質分子を測定できる;ペプチドのような小分子からウィルス等の巨大分子複合体まで、非常に広い範囲の分子量を測定できる;タンパク質のおおよその形状や、分子間の相互作用の強さなどの情報も得られる;などを挙げることができる。
本プロトコールでは、超遠心分析の2つの実験手法「沈降速度法」と「沈降平衡法」について紹介する (2–5)。筆者は沈降平衡法実験を行う場合でもまず沈降速度法実験を行って、タンパク質溶液の性状についての情報をできるだけ収集するようにしている。沈降速度法実験では、夾雑タンパク質の有無や、タンパク質分子の流体力学的均一性がわかる。均一性が高ければ、タンパク質分子のおおよその分子量や形状についての情報が得られる。得られた情報を基にして沈降平衡法実験を行えば、タンパク質の分子量を精密に求めることができる。実験にかかる日数や時間は、実験手法および測定対象のタンパク質の分子量によって異なる。
目的・イントロダクション等
筆者の周囲ではタンパク質の結晶構造解析や NMR 解析が盛んに行われている。超遠心分析は、それらの解析に非常に役立つ情報を提供することができる。例えば、
- 調製されたタンパク質溶液中に変なアグリゲーションはないか?それともタンパク質は均一か?
- 溶液中でそのタンパク質はモノマーか?それとも自己会合している?
- タンパク質が自己会合している場合、平衡定数はどのくらい?
- タンパク質 A とタンパク質 B は結合するか?
- 結晶化したいタンパク質複合体がキチンとできているか?
などの疑問にこたえることができる。
沈降速度法とは、タンパク質溶液を強力な遠心力場に置いた際に、「沈降係数」の大きいタンパク質ほど速く溶媒の中を沈降することを利用して、そのタンパク質溶液の流体力学的均一性と沈降係数の分布を見積もる実験手法である。溶媒中のタンパク質分子が底に向かって沈降を始めると、溶媒中のタンパク質がなくなった部分とまだ存在している部分の間に、「移動境界面」が生じる(図1)。この移動境界面の曲線の形状や時間変化が、タンパク質分子の沈降係数と拡散係数の大きさを反映するため、これを分光学的に観測・記録して、解析に用いる。ここで、繊維状タンパク質や扁平な形状のタンパク質は、同じ分子量の球状タンパク質と比べて拡散係数が小さく、従って遅く沈降する。また、沈降係数と拡散係数(または拡散係数と反比例の関係にある「摩擦係数」)からタンパク質分子の形状に関する情報を得ることができる。
実験にかかる時間は、タンパク質の分子量に依存する。すなわち、分子量が大きいタンパク質ほど速く沈降するので、大きなもの(100 kDa 程度以上)は数時間でデータ収集が終わるが、分子量の小さなタンパク質(10 kDa 程度以下)は一晩以上遠心を続けることがある。
一方、沈降平衡法とは、タンパク質溶液を比較的弱い遠心力場に置き、その結果、溶液中に生じるタンパク質の濃度勾配を測定する実験手法である。特に、タンパク質分子にかかる遠心力、浮力、濃度差に起因する拡散、の3つが釣り合って平衡状態に達した状態を分光学的に測定する。この場合、タンパク質の濃度を回転中心からの距離を横軸にとってプロットすると分子量に依存した指数関数になることが知られており、測定で得られた曲線に理論曲線をカーブフィッティングして分子量を決定することができる(図2)。
実験にかかる時間は速度法実験よりも長い。分子量が大きいタンパク質ほど平衡に達するまでの時間がかかり、100 kDa 程度の分子量のタンパク質では丸1日程度必要である。多くの場合、複数の回転数条件を使ってデータ収集をするため、終了までに数日間を要する。
装置・器具・試薬
- 代表的な超遠心分析機、Beckman Coulter 社の分子間相互作用解析システム XL-A(もしくは XL-I)および付属品(制御用 PC、ローター、サンプルセル、カウンターバランス等)を利用することを想定する。
- 装置に付属してくるサンプル注入用のビニール製チューブとシリンジ、もしくは先の細いチップとピペットマン(各社)
- 装置制御用 PC の他に、データ解析用の PC(Windows)があればなお良い。データ解析用ソフトウェア(SEDNTERP (6)、SEDFIT (7,8) それぞれ
http://www.jphilo.mailway.com/download.htm
http://www.analyticalultracentrifugation.com/default.htm[リンク切れ]
から無料でダウンロード可)を PC にインストールしておく。 - 解析対象のタンパク質溶液と、光学系レファレンスに用いる緩衝液。タンパク質溶液は、この緩衝液で透析されたものが望ましい。
実験手順
沈降速度法実験
- サンプルの用意
- サンプルセルを組み立ててサンプルを入れる
- サンプルセル、ローター、光学系の設置
- テストデータ測定、温度平衡
- 本番データ測定
- 実験終了、サンプル回収
- データ解析
沈降平衡法実験
- サンプルの用意
- サンプルセルを組み立ててサンプルを入れる
- サンプルセル、ローター、光学系の設置
- テストデータ測定
- 本番データ測定
- 実験終了、サンプル回収
- データ解析
実験の詳細
沈降速度法実験
1. サンプルの用意
実験には、測定対象のタンパク質溶液と、光学系レファレンスに用いる緩衝液の2つが必要である。タンパク質溶液は、この緩衝液で透析されたものが望ましい。
タンパク質溶液は、X 線結晶構造解析や NMR 測定に用いられるような、SDS ゲル電気泳動で単一バンドになるくらいの純度のものであれば問題ない。タンパク質の測定には 280 nm の光吸収がよく用いられるが、波長スキャンを行い、ピーク波長で測定するのが望ましい。沈降速度法に用いるタンパク質溶液は、測定波長での吸光度が0.8~1.0程度のものを 400 μL 用意する。試料に余裕があれば、これ以外に吸光度が0.5程度、0.25程度のものも 400 μL ずつ用意する。タンパク溶液濃度が低い場合や、タンパク質が 280 nm にほとんど光吸収を持たない場合は、測定波長に 230 nm を用いることもできる。また、可視光領域の波長も利用可能なので、ヘムタンパク質等の測定に利用できる。
緩衝液は、トリス-HCl やリン酸など一般的なものであれば問題ない(ただし、230 nm などの短波長の光吸収を利用して測定する場合は、トリスや酢酸系緩衝液は避けるべきである)。緩衝液中の塩濃度が重要である。タンパク質分子同士の非特異的な静電相互作用を抑えて、沈降実験に理想的な溶液状況にするために、塩濃度を 0.1~0.2 M 程度にする。また、測定波長周辺に大きな光吸収を持つ物質、例えば ATP が緩衝液に入っていると、光吸収を用いたタンパク質の測定は難しい(他の測定方法を後述する)。DTT やメルカプトエタノール等の還元剤も 280 nm 付近に光吸収があり、時間とともに変化してしまう恐れがあるので緩衝液に入れるのはおすすめしない。TCEP などの 280 nm の光吸収が少ない還元剤が代わりに使われた報告がある。グリセロールを入れても問題はない。ただし、緩衝液の粘性が高くなってタンパク質の沈降が遅くなる。
2. サンプルセルを組み立ててサンプルを入れる
サンプルセルは非常に高価なので丁寧に扱う。沈降速度法では、2つの穴を持つ「ダブルセクターセンターピース」と呼ばれる部品と、「ウィンドウアセンブリ」、専用の「セルハウジング」などが使われる。組み立て部品は非常に多い(図3)。装置マニュアルに従って正確に組み立てる。センターピースとウィンドウアセンブリはぴったりとつかなければならない。間にホコリがはさまると、遠心の際中にサンプルが漏れてしまい、実験は失敗、サンプル回収もできなくなる。エアダスターなどを用いてホコリを吹き飛ばしつつ、サンプルセルを組み立てる。
速度法用ダブルセクターセルには、円筒形セルの側面に小さな穴が2つある。この穴を通してタンパク質溶液 400 μL、緩衝液 420 μL をそれぞれ注入する。装置付属のチューブと 1 ml シリンジ、もしくは先端の細いチップとピペットマンを用いて容積をきちんと測り取る。穴はポリエチレン製の専用フィルムで塞ぎ、その上を小さなねじ式フタで締める。アルコールなどの極性の高い有機溶媒を使用する際には、穴とポリエチレンフィルムの間にテフロンシールを入れると液もれを防ぐのに役立つ。
3. サンプルセル、ローター、光学系の設置
サンプルセルと「カウンターバランス」と呼ばれる基準セルの重さがつり合うように、カウンターバランスの重さを調節する。カウンターバランスは必ずローターの4番穴(50Ti ローターの場合は8番穴)に入れ、その対位置にサンプルセルを入れる。複数のサンプルセルを測定したい場合は、測定する全てのサンプルセルの重さをはかり、重さの差が 0.5 g 未満のもの同士を選んでローターの対位置に入れる。残りのサンプルセルとカウンターバランスの重さを合わせてローターに入れる。
サンプルセルを入れたローターと光学系を装置チャンバー内に設置する。フタを閉じて測定温度を設定後、チャンバー内を真空ポンプ装置で減圧する。
4. テストデータ測定、温度平衡
まず 3,000 rpm 程度の回転数条件で1回だけデータ測定してみて(Single Scan)、パラメータに間違いがないか確認する。パラメータは、ローターのタイプ(4穴 or 8穴)、回転数、時間、温度、データ収集をしたいサンプルセルの番号、実験方法(波長測定 or 速度法実験 or 平衡法実験)、測定範囲(回転中心からの距離 cm)、測定波長(nm)、測定データを格納するディレクトリ情報、使用しているセンターピースの形状、等々である。問題がなければいったんローターの回転を止める。真空ポンプ装置によるチャンバー内減圧は維持する。チャンバー、ローター内の温度が完全に平衡に達するように、チャンバー内温度が設定温度に達してからさらに1時間ほど待った後に本番のデータ測定を開始することが望ましい。超遠心分析の実験では、20℃を標準温度として用いることになっている。
5. 本番データ測定
測定のための最適なローター回転数は沈降係数に依存するが、分子量 50 kDa 以下のタンパク質ではローターの最高回転数を用いるのが普通である(目安は 40,000~60,000 rpm)。測定回転数に達してからデータ収集を開始する(Method Scan)。5~10分に1回スキャンするのが標準的である。全てのタンパク質分子種がセルの底に沈降するまで遠心およびデータ収集を続けるのがよい。100 kDa 以上の大きなものは、回転開始から終了までに数時間かかる。10 kDa に満たない分子量のタンパク質がすべて沈降するまでには10時間以上かかることもある。ペプチドのような小分子はセルの底まで沈降しないので、それ以上沈降しない、というところまで回転とデータ収集を続ける。
6. 実験終了、サンプル回収
データ収集終了後、ローターの回転を止め、拡散ポンプが冷めるのを待ってからチャンバー内の減圧を解除する。測定後のサンプルは、サンプルセル注入穴から9割以上回収できる。
7. データ解析
データ解析の前に、あらかじめタンパク質や緩衝液の物理化学的な性質について情報収集をしておく。超遠心分析のデータ解析には、タンパク質分子に働く浮力や摩擦力が重要なので、タンパク質の偏比容、緩衝液の密度・粘度の数値が必要である。偏比容(partial specific volume, v-bar, 部分比容と呼ばれることもある)(9) は、1 g の溶質を多量の溶媒に溶かした時の溶液の容積増加として定義され、単位は mL/g である。タンパク質の偏比容を精密に測定するためには振動式密度計を使用するが、この観測は手間がかかるため、代わりに構成アミノ酸残基の偏比容の重量平均を計算する。タンパク質の偏比容、緩衝液の密度、粘度の数値は、Laue らが開発したソフトウェア「SEDNTERP」で計算することができる。
沈降速度法では、異なるタンパク質濃度で実験を行って得られたデータはそれぞれ別々に解析を行う。解析のためのソフトウェアにはいろいろなものがあるが、ここでは Schuck らが開発した SEDFIT と呼ばれる Windows 用ソフトを紹介する。SEDFIT ウェブサイト(http://www.analyticalultracentrifugation.com/default.htm[リンク切れ])には、理論的背景、具体的な使用方法、サンプルデータなどがきめ細かく掲載されている。SEDFIT の最大の特徴は、沈降速度法実験データから非常に分解能の高い「沈降係数分布関数、\(\mathrm{c(s)}\)」を得られる所だろう。グラフの横軸は沈降係数 \(\mathrm{s}\)(単位 S、沈降係数 \(\mathrm{s}\) は秒の単位を持つ。10–13秒を Svedberg 単位、S として書き表す。Svedberg は、1920年代に超遠心分析の先駆的な研究を行った人物で、1926年に「分散コロイドに関する研究」でノーベル賞を受賞した)である。縦軸は「沈降係数 \(\mathrm{s}\) と \(\mathrm{s + \Delta s}\) の間に存在する分子種の濃度」と定義された \(\mathrm{c(s)}\) の大きさである(単位は A/S、A は吸光度、S は沈降係数の単位)。\(\mathrm{c(s)}\) のグラフはゲルろ過カラムからのタンパク質溶出パターンに似ている。ただし、ゲルろ過では分子量の大きなタンパク質ほど早く溶出するので、左右が逆になる。大きい \(\mathrm{s}\) 値を持つタンパク質ほど一般的には分子量が大きい。また、ピークの大きさはその成分の存在量(重量濃度に比例)を示す。
\(\mathrm{c(s)}\) グラフの形から読み取れることを何点か挙げる。
- シャープな形のピークが1つだけで、ピークの \(\mathrm{s}\) 値にタンパク質の濃度依存性がほとんどない場合(図4):これはタンパク質の均一性が高く、非常に安定であることを示している。このケースでは、SEDFIT でタンパク質分子の「摩擦比」も求めることができる。摩擦比とは流体力学の用語であるが、タンパク質の形状が真球に比べてどのくらい偏っているかを知る目安となる。通常の球状タンパク質であれば、1.05から1.30程度の摩擦比を示すことが多い。細長い形状や扁平な形状のタンパク質は、これよりも大きな摩擦比を示す。沈降係数 \(\mathrm{s}\)、および摩擦比(または拡散係数)が求まると、タンパク質分子の分子量も求めることができる。SEDFIT では、\(\mathrm{c(s)}\) グラフを「分子量分布、\(\mathrm{c(M)}\)」に計算し直すことができて便利である。
- \(\mathrm{c(s)}\) グラフにピークが複数ある場合(図5、図6):夾雑タンパク質がある、もしくはタンパク質が単量体–多量体平衡などの自己会合状態にあると考えられる。この場合は、複数のタンパク質濃度条件を用いて速度法実験をするべきである。高い濃度のタンパク質を実験に用いると大きなs値を持つ成分が増えるのであれば、可逆的な自己会合・解離の平衡状態にあると考えられる。SEDFIT では \(\mathrm{c(s)}\) から \(\mathrm{c(M)}\) への変換において「全ての分子種の摩擦比が共通である」という仮定をおいているので、SEDFIT だけで正確な分子量を求めることは難しい。正確な分子量は沈降平衡法を用いて求めるべきである。ただし、沈降平衡法で求めることができるのは、個々のピークの分子量ではなくて重量平均の分子量である。(詳細は沈降平衡法実験のデータ解析のページを参照)
- \(\mathrm{c(s)}\) グラフのピークは1つだけだが、形がなだらか、もしくは偏った形である。高い濃度のタンパク質を実験に用いると、ピークの形が変化して \(\mathrm{s}\) 値が大きくなる傾向にある場合(図6):タンパク質が単量体–多量体平衡状態にあり、会合・解離の速度が速いケース(\(\mathrm{koff} > 0.01\text{/sec}\) 程度)と考えられる。この場合も、沈降平衡法を用いてデータ収集を行い、単量体–多量体平衡モデルなどを利用してデータ解析を行うべきである。
沈降平衡法実験
1. サンプルの用意
実験には速度法実験と同様、測定対象のタンパク質溶液と、光学系レファレンスに用いる緩衝液の2つが必要である。タンパク質溶液は、この緩衝液で透析されたものが望ましい。タンパク質溶液は、あらかじめ速度法実験で性質を調べて、できるだけ情報を集めた方が絶対に有利である。夾雑タンパク質の有無、おおよその分子量の情報があるだけで、平衡法実験の計画が立てやすい。
平衡法実験に用いるタンパク質溶液は、6個の穴のあるサンプルセルを用いる場合、測定波長での吸光度が0.15、0.25、0.50程度のものを 110 μL ずつ用意する。ダブルセクターセルを用いる場合は、同じ程度の吸光度のタンパク質溶液を 120 μL 用意する。
緩衝液は、速度法実験と同様、トリス-HCl やリン酸など一般的なものであれば問題ない。緩衝液条件は速度法実験のものとそろえた方がもちろん良い。
2. サンプルセルを組み立ててサンプルを入れる
平衡法実験で6個の穴のあるサンプルセルを用いる場合は、1つのセルで3つの異なる初濃度を測定できる(図7)。タンパク質溶液(110 μL)と緩衝液(120 μL)を隣り合わせるようにセルに入れる。平衡法でダブルセクターセルを用いる場合は、タンパク質溶液と緩衝液をそれぞれ 120 μL および 140 μL 注入する。
3. サンプルセル、ローター、光学系の設置
サンプルセルと「カウンターバランス」と呼ばれる基準セルの重さがつり合うように、カウンターバランスの重さを調節する。カウンターバランスは必ずローターの4番穴(50Ti ローターの場合は8番穴)に入れ、その対位置にサンプルセルを入れる。複数のサンプルセルを測定したい場合は、測定する全てのサンプルセルの重さをはかり、重さの差が 0.5 g 未満のもの同士を選んでローターの対位置に入れる。残りのサンプルセルとカウンターバランスの重さを合わせてローターに入れる。サンプルセルを入れたローターと光学系を装置チャンバー内に設置する。フタを閉じて測定温度を設定後、チャンバー内を真空ポンプ装置で減圧する。
4. テストデータ測定
まず 3,000 rpm 程度の回転数条件で1回だけデータ測定してみて(Single Scan)、パラメータに間違いがないか確認する。パラメータは、ローターのタイプ(4穴 or 8穴)、回転数、時間、温度、データ収集をしたいサンプルセルの番号、実験方法(波長測定 or 速度法実験 or 平衡法実験)、測定範囲(回転中心からの距離 cm)、測定波長(nm)、測定データを格納するディレクトリ情報、使用しているセンターピースの形状、等々である。
5. 本番データ測定、データ収集
データ測定のパラメータを決める。測定用の最適なローター回転数は、タンパク質の沈降係数と拡散係数によって異なる。このため、あらかじめ速度法実験を行ってタンパク質のおおよその分子量または沈降係数を求めておくのがよい。Beckman Coulter 社から最適なローター回転数とタンパク質分子量の関係についてのグラフが提供されているので、これを参考にして回転数を決める。この回転数より30%程度多い回転数、30%程度少ない回転数でもデータ収集する。また最後に、セルのベースライン情報を得るために、大きな回転数でタンパク質を緩衝液の底に沈降させて緩衝液だけの状態にしてデータ収集を行う。(例:分子量 60 kDa 程度のタンパク質を測定する場合、7,000、10,000、13,000、30,000 rpm の4つの回転数でデータ収集を行う)一連のデータ収集を自動で行うようにプログラムが組める。小さい回転数から順番にデータ収集を行う。1つの回転数につき、タンパク質濃度勾配が平衡に達するまで12~48時間かかる。かかるおおよその時間(秒)は \(\text{(solution column height)}^{2}/\mathrm{D}\)(solution column height はタンパク質溶液の底から水面までの長さで単位は cm、D はタンパク質の拡散定数で単位は cm2/s)(10) で推定できるが、粘性の高い緩衝液が使われている場合は、この推定時間よりもさらに長時間かかる。全ての測定が終了するまでに数日間かかる。各回転数で平衡に達したかどうかは、数時間おきにデータ収集を行って、得られた濃度勾配曲線を重ねてプロットして、完全に重なることを見ることによって確認できる。SEDFIT の Option の1つに、平衡法実験で系が平衡に達したかどうかを判断する機能があるので、確認に利用できる。(SEDFIT のメニューのOptions → Loading Options → Test Approach to Equilibrium)
6. 実験終了、サンプル回収
データ収集終了後、ローターの回転を止め、チャンバー内の減圧を解除する。測定後のサンプルは、サンプルセル注入穴から9割以上回収できる。
7. データ解析
沈降平衡法で得られたデータは、XL-A(XL-I)装置に添付されているソフトウェアで解析されることが多い(図8)。タンパク質の3つの初濃度、ローター回転数各3つから合計9個のタンパク質濃度勾配曲線が得られる。9個の実験曲線をまとめて理論曲線でフィッティングする。これは「グローバルフィッティング」と呼ばれる。筆者が解析を行う際には、実験で得られたセルのベースライン情報を使い、ベースラインの値を最小二乗法で浮動させない。
グローバルフィッティングで大事なのが、どのような「理論モデル」を用いるか? である。沈降速度法で得られた情報が、モデル選びの際に重要な役割を果たす。速度法でタンパク質の均一性が高いとわかれば、最も単純な「単一成分モデル」を選べばよい。最小二乗法で浮動させるパラメータはタンパク質の分子量だけである。実験曲線を全てフィッティングできる分子量を、高い精度で求めることができる。タンパク質が単量体–多量体平衡にあると見当がつけば、「単量体–二量体平衡モデル」や「単量体–三量体平衡モデル」等を選ぶ。これらの場合は、タンパク質の分子量と結合定数が浮動パラメータとなる。最も単純なモデルから始めて、必要に応じてより複雑なモデルを試みるのがよい。こうして、実験曲線を最も良くフィッティングできるモデルが妥当ということになる。平衡モデルでは結合定数をパラメータとしてフィッティングを行うので、この解析法でタンパク質の分子量のみならず、相互作用の強さの情報も得ることができる。
工夫とコツ
インターネットやメーリングリストを利用しよう
インターネット上では、超遠心分析に関する意見・情報交換が行われている。以下に代表的なサイトを挙げる。メーリングリストの過去ログ等も閲覧可能なので、困ったことや疑問があればまず検索してみることをお勧めする。
http://www.bbri.org/RASMB/rasmb.html
RASMB 分析超遠心討論グループメーリングリスト、ソフトウェアアーカイブ、FAQ など(英語)
http://bilbo.bio.purdue.edu/mailman/listinfo/sedfit[リンク切れ]
SEDFIT ユーザーグループメーリングリスト(英語)
http://www.farisaka.bio.titech.ac.jp/aucforum/aucclub.html[リンク切れ]
超遠心分析クラブインターネットフォーラムメーリングリスト(日本語)
データ解析用ソフトウェアのインストール
残念ながら SEDNTERP のインストーラーは日本語 WindowsXP に対応していない。裏技的インストール方法が超遠心分析クラブインターネットフォーラム(http://www.farisaka.bio.titech.ac.jp/aucforum/aucclub.html[リンク切れ])で触れられている。筆者も日本語 WindowsXP(SP2) への SEDNTERP インストールに挑戦してみたところ、上記の裏技とほぼ同じ方法で成功した。インストール用ファイルを解凍するためのソフトウェアは WinZip に限らず、一般的なものでも使えるようである。Admin 権限を持つユーザーでログインしないと SEDNTERP を使えなかった。2008年4月現在、SEDNTERP の最新版は version 1.09(9/3/2006) with database revision 1.2 である。
SEDFIT は、まず実行ファイルをダウンロードする。また、C: ドライブ直下に C:\sedfit フォルダを作成して、実行ファイルをそのフォルダ中に入れておけば使える。2008年4月現在、SEDFIT の最新版は version 11.3b である。ひんぱんにバージョンアップされ、その度に bug fix や新しい機能が追加されている。
いずれのソフトウェアも大したマシンスペックは必要ない。SEDFIT は CPU clock 500 MHz, 256 MB RAM が最低限必要で、OS は Windows NT, 95, 98, 2000, XP で動作するそうである。筆者が少し試したところ、Vista 上でも動作するように見える。SEDFIT の最新の機能を使いこなしたり、サクサクした計算をお好みの方はハイスペック PC 利用がおすすめ。
上記、Windows 用ソフトばかりで Mac な方々は悲しんでおられるかもしれない。IntelMac とエミュレータを利用して Windows 環境を用意し、そこで SEDFIT を使用している方がいるようだ。
SEDFIT 以外にもさまざまなデータ解析用ソフトウェアがある。
Dr.Borries Demeler の UltraScan (for Linux, Unix, Windows, MacOS-X)
Van Holde-Weischet法を使った速度法実験データ解析など。
http://www.ultrascan.uthscsa.edu/
http://cauma.uthscsa.edu/
Dr. Walter Stafford の SEDANAL (for Windows), dcdt (for Windows, Mac)
http://www.bbri.org/faculty/stafford/AUCRL.html[リンク切れ]
Dr. John Philo の SVEDBERG, DCDT+ (for Windows)
http://www.jphilo.mailway.com/
Dr. Dmitry Veprintsev の UltraSpin (for Windows)
平衡法実験データ解析用フリーウェア。
http://ultraspin.mrc-cpe.cam.ac.uk/[リンク切れ]
280 nmの光吸収が使えない!(濃いタンパク質サンプルを測りたい、タンパク質が光吸収を持たない、緩衝液に ATP を入れたい、など)そんな場合は…
緩衝液に ATP などの紫外光を吸収する物質が含まれている場合、また非常に濃いタンパク質サンプル、逆にタンパク質がほとんど光吸収を持たない場合は、光吸収を利用したタンパク質の測定はできない。Beckman Coulter 社の XL-I は通常の吸収光学測定系の他に、「レイリー光学干渉計」を付属しているので、このようなサンプルでも実験が可能である。レイリー光学干渉計(Rayleigh interference optical system)とは、物質の屈折率変化を利用して気体や液体の動きを可視化するための光学装置である。Beckman Coulter 社の XL-I は、これを溶液中のタンパク質の動きを可視化するために応用している。
溶液の屈折率は溶質の重量濃度に依存するので、タンパク質がほとんど光吸収を持たなくてもある程度のタンパク濃度があればレイリー干渉計で検出可能である。溶液中に ATP などの物質が含まれていても、その重量濃度はタンパク質のものに比べれば小さいので影響は少ない。また、レイリー干渉計で測定されるデータは非常に S/N 比が良く、非常に濃い濃度のタンパク質溶液でも測定可能である。
ただし、測定用サンプルセルのウィンドウアセンブリーには、コルツ製ウィンドウの代わりにサファイア製ウィンドウを用いる。コルツ製ウィンドウは遠心による圧力で微妙に歪んでしまい、レイリー干渉計で検出されてしまうからである。このため、この測定法を用いる場合はより歪みが少ないサファイア製ウィンドウを用いる。
SAXS とタッグを組む
超遠心分析法は、X 線小角散乱(SAXS)と組み合わせることによって、タンパク質複合体などの構造情報をより詳しく議論できる。SAXS を利用するとタンパク質の低分解能溶液構造を解析できるが、サンプルに制限がある。きれいな単分散を示す均一性の高いタンパク質溶液でなければ SAXS データ解析は難しい。超遠心分析速度法実験でタンパク質の均一性を確認できたサンプルは、SAXS 測定・解析がうまくいくことが多い。あらかじめサンプルの良否や緩衝液条件を検討しておくことにより、貴重なシンクロトロン SAXS ビームタイムを有効に使える。
超遠心分析が、ヒト転写因子 TFIIE 複合体の分子量決定、およびおおよその形状の推定に使われた例を紹介する (11)。TFIIE はαサブユニット(50 kDa)とβサブユニット(35 kDa)か らできている。超遠心分析、ESI-MS(エレクトロスプレーイオン化質量分析)、そして SAXS の3つの手法を用いて TFIIE の分子量および分子形状を見積もる試みがなされた。沈降速度法実験から、TFIIE は非常に安定で、α1β1ヘテロダイマーに相当する分子量を持ち、かつ、2.1という非常に大きな摩擦比を示すことがわかった。この大きな摩擦比は、TFIIE が非常に細長い形状、もしくは扁平な形状を持つことを示す。計算から、細長い葉巻型楕円形(流体力学モデル)に近似すると、長辺と短辺の長さの比15 : 1程度のものが、摩擦比2.1を示すことがわかったので、この摩擦比がいかに特異な数値であるかがわかる。超遠心分析の結果は、ESI-MS、SAXS 低分解能分子構造モデルの結果と一致したので、TFIIE はαサブユニットとβサブユニットそれぞれ1つずつからできたα1β1ヘテロダイマーで、非常に細長い全体構造を持つと結論付けられた。
文献
- Lebowitz, J. et al., Protein Science, 11, 2067–2079 (2002)
- 有坂文雄, 蛋白質核酸酵素, 43(13), 2024–2032 (1998)
- 有坂文雄, 蛋白質核酸酵素, 43(14), 2145–2152 (1998)
- 有坂文雄, 蛋白質核酸酵素, 43(15), 2238–2244 (1998)
- 有坂文雄, 蛋白質核酸酵素, 44(1), 82–91 (1999)
- Laue, T.M. et al. : Analytical Ultracentrifugation in Biochemistry and Polymer Science (eds. Harding, S.E., Rowe, A.J., & Horton, L.C.), pp90–125, Royal Society of Chemistry, Cambridge (1992)
筆者註、本文中の式や表に誤植がある。SEDNTERP ソフトウェア中のヘルプファイルの中では訂正されている。 - Schuck, P., Biophycical Journal, 78(3), 1606–1619 (2000)
- Schuck, P., et al., Biophycical Journal, 82(2), 1096–1111 (2002)
- 生化学事典第3版、東京化学同人 (1998)
- van Holde, K.E., & Baldwin, R.L., J.Phys.Chem., 62(6), 734–745 (1958)
- Ito, Y. et al., Proteins, 61(3), 633–641(2005)
2021年8月13日追記
2008年に「超遠心分析を用いた蛋白質の分子量の決定」を発表して以来、解析用ソフトウェアがアップデートされ続けている。今回は、現時点(2021年8月)での主なソフトウェアの最新情報を提供したい。
緩衝液条件、蛋白質の偏比容計算などに使われるソフトウェア SEDNTERP
SEDNTERP 3 紹介ページ:
http://www.jphilo.mailway.com/sednterp.htm
ダウンロード先:
http://www.jphilo.mailway.com/download.htm
最新版は Version 3.0.3 で、リリース日は2021年3月14日。インストーラー(sednterp3_setup.exe)は Windows 8, 10 環境で動作する。
超遠心分析データ解析用ソフトウェア SEDFIT
http://www.analyticalultracentrifugation.com/default.htm[リンク切れ]
最新版は Version 16.1c で、リリース日は2018年10月23日。Windows 環境で動作する。旧版を含めて、SEDFIT はマルチコアプロセッサでの計算に対応している。SEDFIT のメニュー中、Options → Size-Distribution Options → Compute with multiple threads の該当箇所を適切に設定することで、計算速度を高速化することができる。
グラフ作成ソフトウェア GUSSI
https://www.utsouthwestern.edu/labs/mbr/software/
SEDFIT、SEDPHAT などのソフトウェアで解析した結果やグラフを、論文発表に使えるクオリティの図にすることができる。最新版は Version 1.4.2 で、リリース日は2018年7月24日。Windows 環境で動作する。インストール方法が以下のリンクに記されている。
https://www.utsouthwestern.edu/labs/mbr/assets/README.txt
正しくインストールすると、SEDFIT や SEDPHAT のメニューに「Plot」が新たに加わる。SEDFIT などで解析を一通り行ってから、Plot 中のリンクをクリックすることで GUSSI が起動し、\(\mathrm{c(s)}\) グラフなどの図を作成することができる。GUSSI を使ってグラフなどのデータを保存しておけば、GUSSI 単独で起動して保存データを読み込み、好みに合わせてグラフの重ね合わせ・線色・軸スケールなどを調節できる。
蛋白質科学会プロトコール編集委員会による追記
尚、本プロトコールに記載されている下記のサイトのリンク切れを2022年7月時点で蛋白質科学会プロトコール編集委員会において確認している:
- http://www.analyticalultracentrifugation.com/default.htm
- http://bilbo.bio.purdue.edu/mailman/listinfo/sedfit
- http://www.farisaka.bio.titech.ac.jp/aucforum/aucclub.html
- http://www.bbri.org/faculty/stafford/AUCRL.html
- http://ultraspin.mrc-cpe.cam.ac.uk/
-
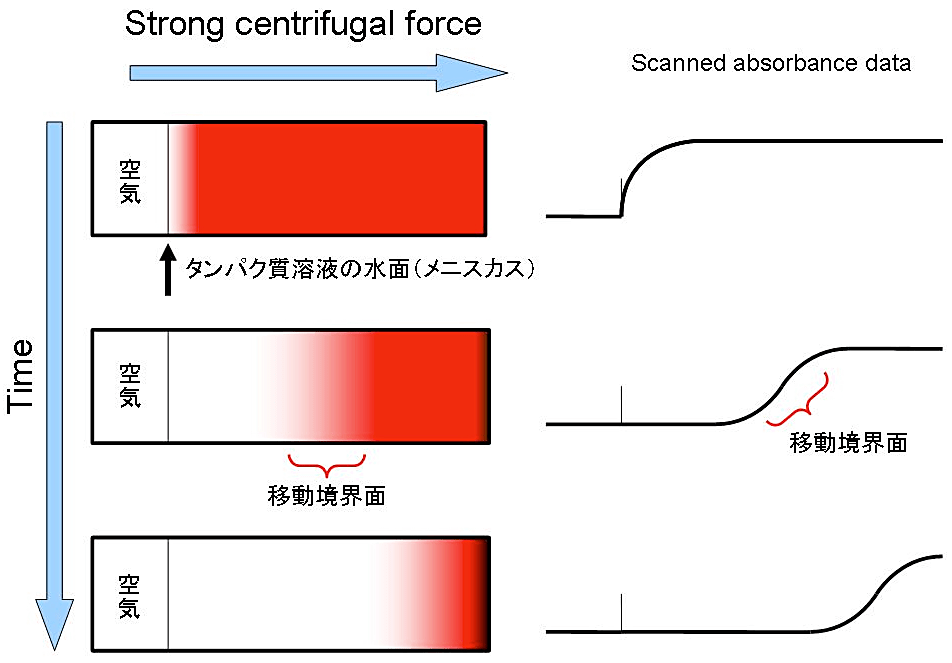
図1:超遠心分析沈降速度法実験の模式図
細長いセルの中にタンパク質溶液を入れて強い遠心力をかけると、溶媒中のタンパク質分子が底に向かって沈降を始める。溶媒中のタンパク質がなくなった部分とまだ存在している部分の間に、「移動境界面」が生じる。この移動境界面の曲線の形状や時間変化が、タンパク質分子の沈降係数と拡散係数の大きさを反映しているので、これを分光学的に観測・記録して、解析に用いる。図中、左側がタンパク質の沈降の様子の模式図で、わかりやすくするために赤色のタンパク質を想定した。実際にはほとんどのタンパク質は無色で、測定には 280 nm の光吸収などが利用される。右側が観測された光吸収のグラフ模式図である。縦軸は吸光度、横軸は回転中心からの距離を示す。移動境界面は時間と共に右(遠心力の方向)に動いていく。最終的にタンパク質は溶液の底に濃縮されることになるが、その濃縮の様子は計算には使われない。 -
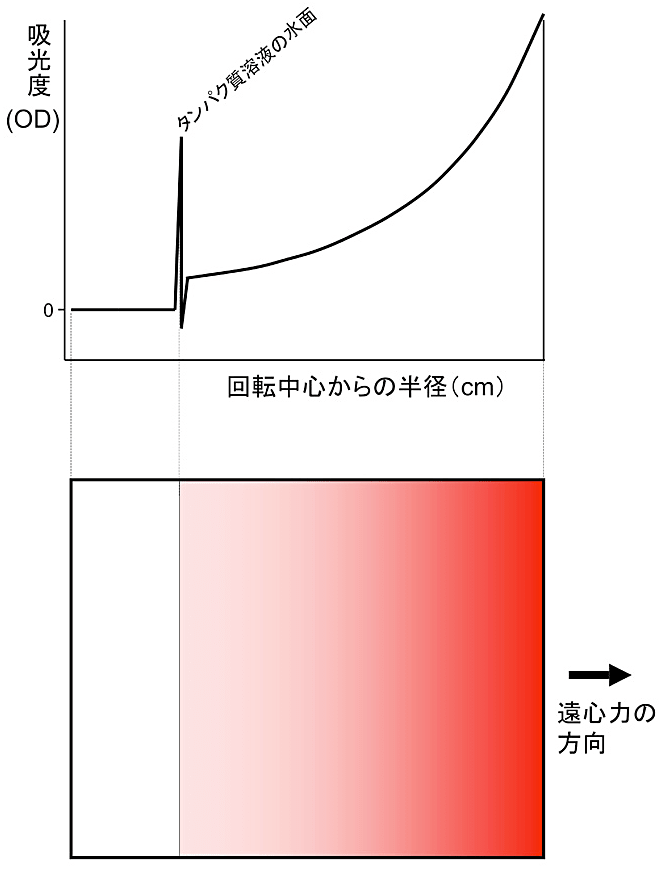
図2:超遠心分析沈降平衡法実験の模式図
沈降平衡法では、タンパク質溶液を比較的弱い遠心力場に置き、その結果、溶液中に生じるタンパク質の濃度勾配を測定する実験手法である。図中では、わかりやすくするために赤色のタンパク質を想定した。タンパク質分子にかかる遠心力、浮力、濃度差に起因する拡散、の3つが釣り合って平衡状態に達した状態を分光学的に測定する。タンパク質の濃度を回転中心からの距離を横軸にとってプロットすると指数関数になることが知られており、測定で得られた曲線に理論曲線をカーブフィッティングして分子量を決定することができる。 -

図3:超遠心分析測定用サンプルセルの部品
速度法実験用サンプルセルの部品をすべて並べてみた。右側2列目の黒くて丸くて2つの穴を持つ部品が「ダブルセクターセンターピース」と呼ばれる。左側半分の部品群は「ウィンドウアセンブリー」のためのもの。 -
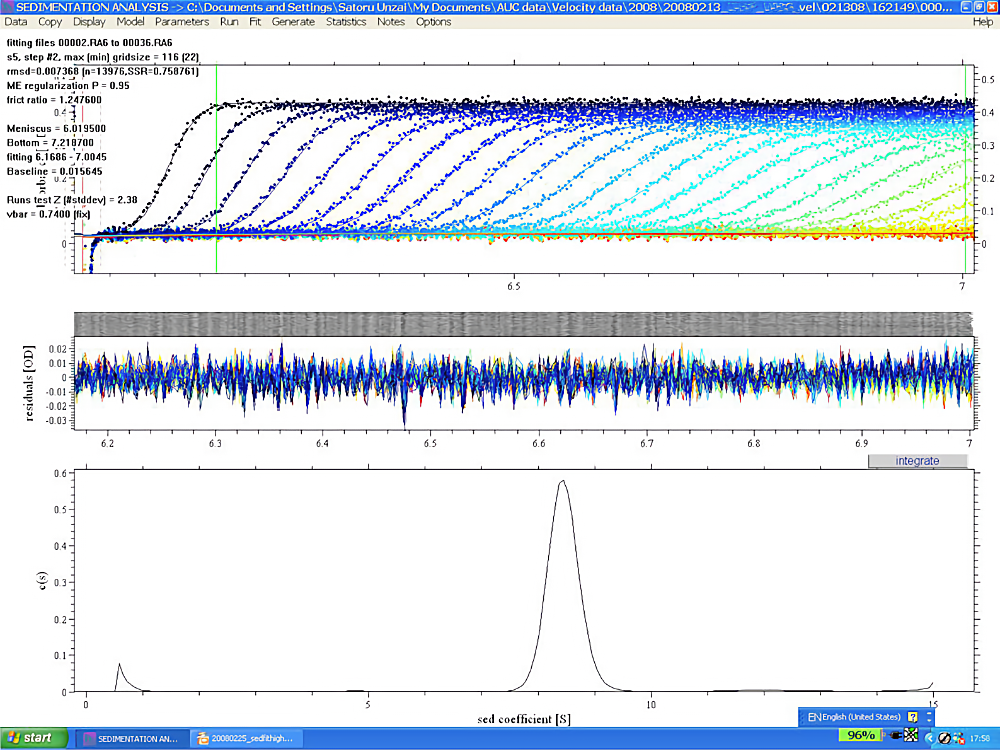
図4:SEDFIT 解析例その1
分子量約 180 kDa のタンパク質複合体について沈降速度法実験を行い、SEDFIT を用いて解析した。上のブロックの小さなドットで描かれた曲線が観測された移動境界面の様子である。縦軸は吸光度、横軸は回転中心からの距離(cm)を示す。50,000 krpm でサンプルを遠心し、5分ごとに1回スキャンしたものを35回分、約3時間分のデータを重ねてプロットしてある。曲線ごとに色分けされているが、見やすくするためのものであまり意味はない。実測値である小さなドットの間を通るように曲線が描かれており、これがカーブフィッティングの結果得られた best fit 理論曲線である。まん中のブロックのグラフは、実測曲線と理論曲線の残差を示しており、35本のデータの残差をすべて重ねてプロットしてある。縦軸は吸光度、横軸は回転中心からの距離(cm)を示す。残差はごく小さく、端から端まで一様なので、実測曲線と理論曲線は非常に良く一致していると言える。下のブロックのグラフが、解析結果の \(\mathrm{c(s)}\) グラフである。縦軸は \(\mathrm{c(s)}\) の大きさ(単位 A/S)、横軸は沈降係数 \(\mathrm{s}\)(単位 S)を示す。\(\mathrm{c(s)}\) グラフは、タンパク質はほぼ均一で沈降係数約 8.4 S を持つことを示す。このタンパク質について、異なるタンパク質濃度での沈降速度法実験も行ったが、ほぼ同じ結果が得られた。これらの結果から、タンパク質溶液の均一性を証明することができた。 -
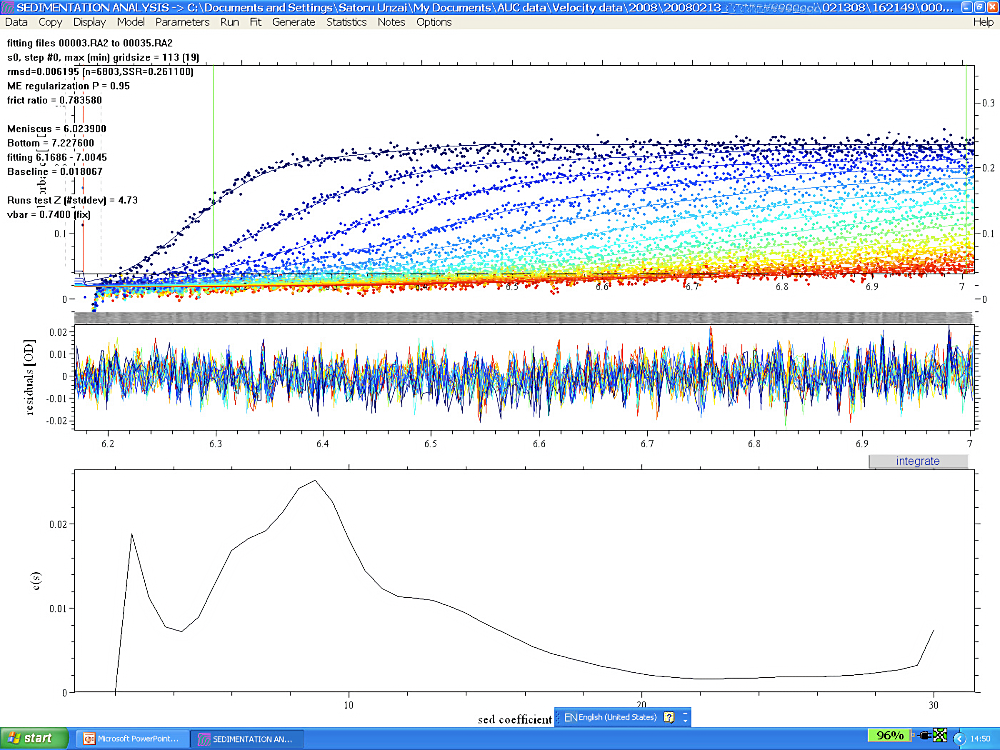
図5:SEDFIT 解析例その2
図4で例に挙げたタンパク質複合体のミュータントを作成し、沈降速度法実験を行って SEDFIT で解析した。\(\mathrm{c(s)}\) グラフには複数のピークがあり、沈降係数の小さなものから大きなものまで、様々な成分があることを示している。異なるタンパク質濃度で同様の実験を行ったところ、ほぼ同様の結果が得られたので、このミュータントタンパク質は非常に均一性が低い;非可逆な会合が起こっている;複合体形成できなかったフリーのサブユニットも含まれる;などが考えられた。 -
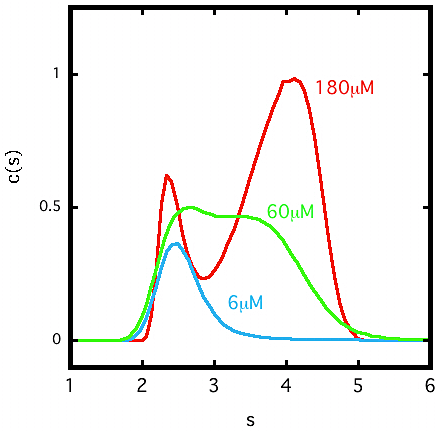
図6:SEDFIT 解析例その3
あるタンパク質の沈降速度法実験を異なるタンパク質濃度条件で3つ行い、SEDFIT で解析をしたところ、それぞれの実験で \(\mathrm{c(s)}\) グラフの形が異なった。\(\mathrm{c(s)}\) グラフの右肩にタンパク質の濃度を示した。縦軸は \(\mathrm{c(s)}\) の大きさ、横軸は沈降係数 \(\mathrm{s}\) を示す。ただし、\(\mathrm{c(s)}\) の縦軸方向の大きさは適度に正規化してある。タンパク質濃度が低い 6 μM では \(\mathrm{c(s)}\) グラフのピークは 2.3 S 程度であるが、60 μM の濃度では右側に shoulder があらわれる。さらに 180 μM の濃度では 4.0 S のピークがはっきりとあらわれる。これらの結果からこのタンパク質は、可逆的な自己会合状態にあると考えられた。分子同士の結合・解離が速い「動的平衡」にある系では、\(\mathrm{c(s)}\) にあらわれるピークは重量平均化されてしまっていて、実在する分子種のピークを表すとは限らない。 -

図7:超遠心分析測定用6穴サンプルセル
専用セルハウジングに入った6穴センターピースの様子を示す。左側の3つの穴に、異なる3つの濃度のタンパク質溶液を 110 μL ずつ、右側の3つの穴にレファレンスに用いられる緩衝液を 120 μL ずつ入れることになる。 -
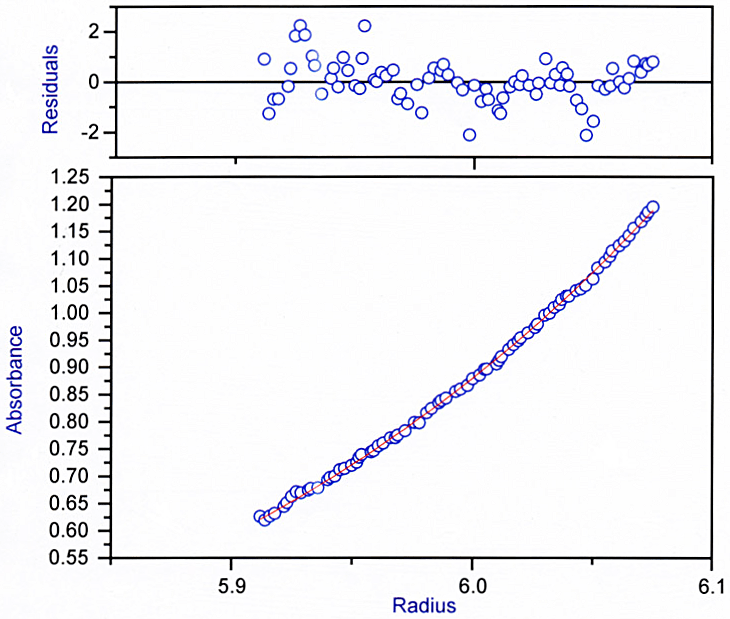
図8:沈降平衡法データ解析例
あるタンパク質について沈降平衡法実験を行い、A280 nm で測定された濃度勾配の様子(下のブロック)を示す。縦軸は吸光度、横軸は回転中心からの距離(cm)、である。青丸が実測データであり、中心付近を通っている赤い線は「single species model」を利用してカーブフィッティングを行い、その結果得られた best fit 理論曲線である。上のブロックは実測曲線と理論曲線の残差である(ただし、残差の縦軸は単純な「実測曲線マイナス理論曲線」の値ではない)。残差のドットは ±0 横線を中心にランダムに分布しているため、実測曲線と理論曲線は良く一致していると言える。


