Dicer-2 とパートナータンパク質による小分子 RNA 産生、受け渡しのスナップショット
東京大学・大学院理学系研究科- 山口そのみ, 蛋白質科学会アーカイブ, 15, Essay 11 (2022)
- https://www.pssj.jp/archives/essay/Es_11/Es_11.html
はじめに
この度は、第20回日本蛋白質科学会年会にて若手奨励賞優秀賞に選んでいただき、誠にありがとうございました。また、久々のオンサイトでの若手奨励賞シンポジウムを開催していただき、ご尽力いただいた先生方、学会スタッフの皆様に感謝申し上げます。
真核生物では、microRNA(miRNA)や低分子干渉 RNA(siRNA)などの20塩基ほどの小分子 RNA が Argonaute タンパク質と RNA induced silencing complex(RISC)と呼ばれるエフェクター複合体を形成し、相補的な塩基配列をもつ標的遺伝子の発現を制御しています。この現象は RNA サイレンシングとよばれ、ショウジョウバエなどの昆虫や植物においては自然免疫機構として、ヒトなどの哺乳類で遺伝子発現抑制機構としてはたらきます (1)。Dicer は RNA サイレンシングで中心的な役割を果たす RNase III 酵素であり、小分子 RNA の生合成と生物種によっては小分子 RNA の Argonaute への受け渡しを行います。近年、クライオ電子顕微鏡による単粒子解析により、Dicer の分子機構が明らかになってきました。本アーカイブでは若手奨励賞シンポジウムで紹介しきれなかった Dicer-2‒R2D2 の構造が解けるまでの過程と筆者らの論文と Su らによって解かれた Dicer-2‒Loqs-PD‒基質二本鎖 RNA の構造について紹介したいと思います。
Dicer-2 の役割と Dicer タンパク質の立体構造解析
RNA サイレンシングはショウジョウバエをモデル生物として研究が進められていました。ショウジョウバエには Dicer-1 と Dicer-2 という2種類の Dicer 酵素があり、Dicer-1 は短いヘアピン miRNA 前駆体を切断して二本鎖 miRNA を産生し、Dicer-2 は長い二本鎖RNA 基質を切断して二本鎖 siRNA を産生します。その後、Argonaute1 はmiRNA と RISC を形成しメッセンジャー RNA を翻訳抑制する一方、Argonaute2(Ago2)は siRNA とRISC を形成しウイルスやトランスポゾン由来の標的 RNA を切断します (2,3)。
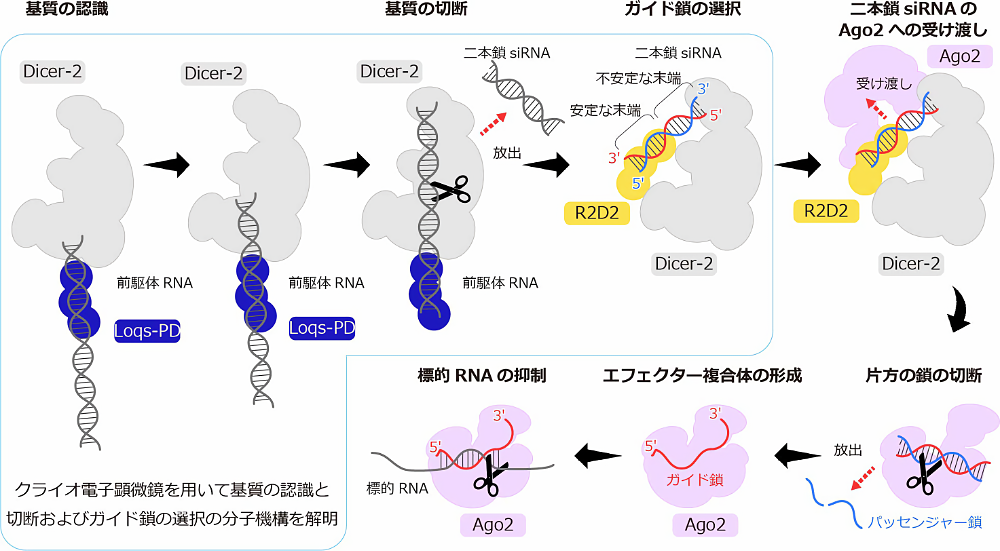
Su らと筆者らは siRNA 経路ではたらく Dicer-2 に着目しました(図1)。Dicer-2 はパートナータンパク質 Loqs-PD と結合し基質の切断を行い、二本鎖 siRNA を産生します (4)。二本鎖 siRNA は Dicer-2‒Loqs-PD から放出され、Dicer-2 と別のパートナータンパク質 R2D2 と再結合します。Dicer-2‒R2D2 は二本鎖 siRNA と一方向で結合し、Ago2 に受け渡すことが知られています (5‒7)。具体的には二本鎖 siRNA の熱力学的により安定な 5′ 末端は R2D2 付近に位置し、より不安定なもう一方の 5′ 末端は Dicer-2 付近に位置しています (8)。二本鎖 siRNA を受け渡された Ago2 は、熱力学的に不安定な 5′ 末端を持つ siRNA 鎖をガイド鎖として使用し、もう一方の鎖をパッセンジャー鎖として放出します (9,10)。このように、Dicer-2‒R2D2 はガイド鎖を選んで Ago2 に受け渡すことで、Ago2 の適切なガイド鎖の取り込みを促進していることが示唆されてきました。
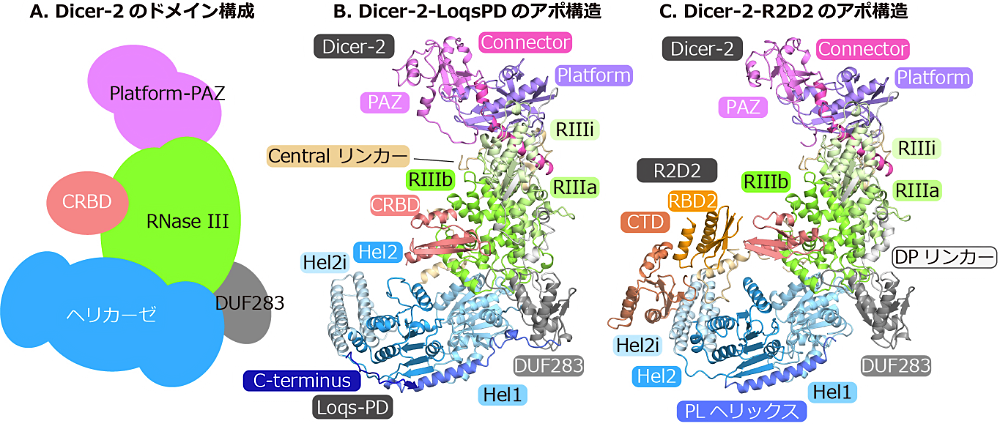
Dicer は主に、アミノ末端(N 末端)のヘリカーゼドメイン、Platform-PAZ ドメイン、RNase III ドメイン、カルボキシ末端(C 末端)の RNA 結合ドメイン(CRBD)から構成されています(図2A)。2006年にヘリカーゼドメインを持たない Giardia intestinalis Dicer の結晶構造が報告され、構造中の PAZ ドメインの RNA 結合ポケットと RNase III ドメインの活性部位との距離は切断産物の長さと一致していました (11)。このことから Dicer は基質 RNA の末端から特定の距離を測定して切断する分子定規であることが提唱されました。2018年にはヒト Dicer‒パートナータンパク TRBP‒miRNA 前駆体、ショウジョウバエ Dicer-2 の基質 RNA の密度マップ(それぞれ4.7 Åと6.8 Å分解能)が報告され、基質 RNA の認識機構が明らかになりました (12)。2021年には植物 Dicer である DCL1、DCL3 と基質 RNA との立体構造が報告され、基質 RNA の切断機構が明らかになりました (13,14)。しかし、Dicer がどのように基質 RNA をRNase III の活性部位に配置するのか、どのように二本鎖 siRNA の熱力学的非対称性を感知し Argonaute へ一定の方向で取り込ませるかは謎のままでした。
Dicer-2‒R2D2 の立体構造が明らかになるまで
このプロジェクトは筆者が修士1年のときから始まりました。Dicer-2‒R2D2 の分子機構の解明は長年の課題となっていましたが、高分解能構造はドメイン単位ですら報告されていませんでした。主な理由として、R2D2 は単体でアグリゲーションを起こすため精製が困難であったことがあげられます。筆者らは Dicer-2‒R2D2 の発現に昆虫細胞内での共発現系を用い、タグを2種類用いた二段階精製で 1 : 1 のモル比で結合した Dicer-2‒R2D2 を調製するように工夫しました。そして Dicer-2‒R2D2‒二本鎖 siRNA 複合体を研究室でよく使用されている QUANTIFOIL grid(R1.2/1.3, Au, 300 mesh)を使い、クライオ電子顕微鏡を用いて単粒子解析を行ったところ、特定の方位を持つ粒子しか観察されず、構造決定が困難でした。筆者らはグリッドの種類を検討し、氷が少し厚めになる QUANTIFOIL grid(R1.0/1.0, Au, 300 mesh)で撮影することにより、様々な方位の粒子を撮影することに成功し、Dicer-2‒R2D2 アポ状態と二本鎖 siRNA と結合した状態に関して3.3 Åと高分解能での構造決定に成功しました (15)。また、同時期に Su らは表面にグラフェンを蒸着させたグリッドを用い、Dicer-2‒Loqs-PD のアポ状態と基質二本鎖 RNA と結合した複数の状態に関して3.3 Å‒4.6 Åの密度マップを得ることに成功しました (16)。グリッドの検討に関して、平泉さんによる蛋白質科学会アーカイブがございますので、そちらも併せてお読みください (17)。
Dicer-2‒Loqs-PD、Dicer-2‒R2D2 のドメイン構成と全体構造
Dicer-2 は5つのドメイン、ヘリカーゼドメイン、DUF283 ドメイン、Platform-PAZ ドメイン、二つの RNase III ドメイン(RIIIa と RIIIb)、CRBD から構成されています(図2A)。また、Dicer-2‒Loqs-PD、Dicer-2‒R2D2 の立体構造より、DUF283 と Platform ドメインをつなぐ DUF283-Platform リンカー(DP リンカー)、RIIIa ドメイン内に挿入されたα-helical ドメイン(RIIIi)と、PAZ とRIIIa ドメインをつなぐCentral リンカー(Su らの論文では PR リンカーと表記されている)の存在が明らかになりました。ヘリカーゼドメインは、Hel1、Hel2、Hel2i ドメインと、Princer-like ヘリックス(PL ヘリックス)から構成されています。Platform-PAZ ドメインは、Platform、PAZ ドメインとコネクターヘリックスから構成されており、基質 RNA の 5′ 末端と 3′ 末端を認識する RNA 結合ポケットがあります。RNase III ドメインは RIIIa、RIIIb ドメインから構成され、これらは分子内二量体を形成し、中央の RNase III 活性部位を形成しています(図2B, C)。
R2D2 は N 末端側の2つの RNA 結合ドメイン(RBD1、RBD2)、RBD と似たフォールドを持つ C 末端ドメイン(CTD)から構成されます。一方、Loqs-PD は R2D2 同様 RBD1、RBD2 を持ちながら、RBD フォールドをとらない25残基程度からなる C 末端を持ちます。Loqs-PD の C 末端は Hel2 ドメインと相互作用する(図2B)一方で、R2D2 では CTD と RBD2 がそれぞれ Hel2i ドメインおよび Central リンカーと相互作用していました(図2C)。また Loqs-PD の RBD1、RBD2 および R2D2 の RBD1 の密度は観察されず、柔軟な構造をとっていることが示唆されました。
Dicer-2‒Loqs-PD、R2D2 による siRNA 経路のスナップショット
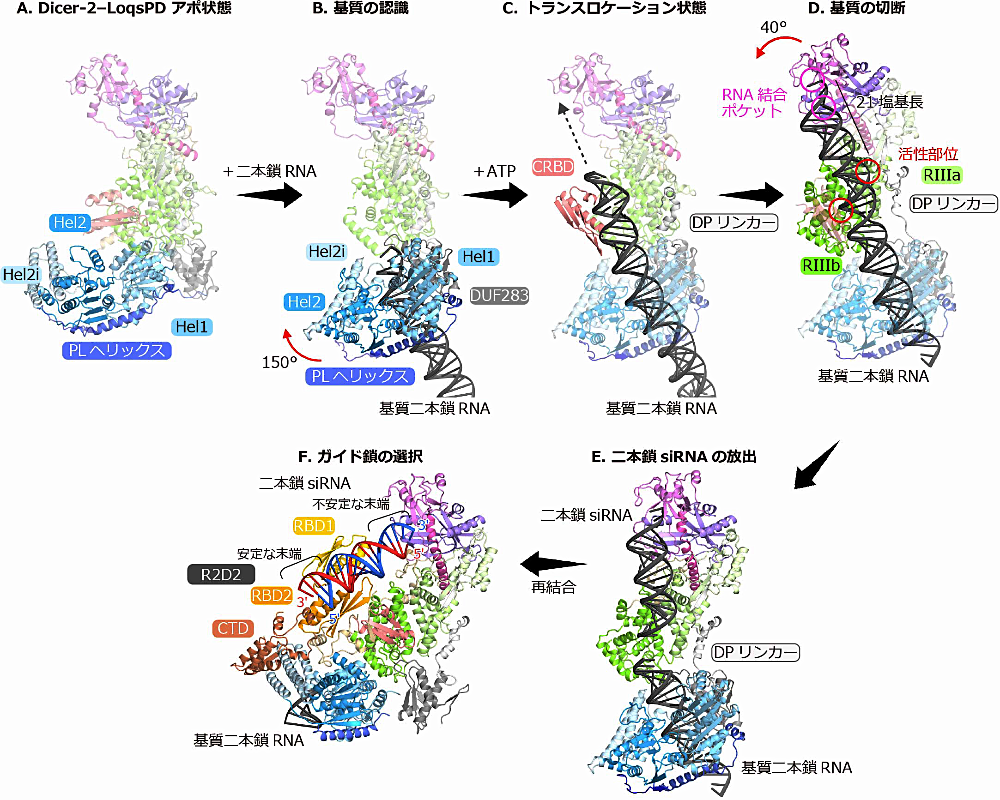
Dicer-2 は基質 RNA をヘリカーゼドメインによって認識することが知られています。Dicer-2‒Loqs-PD は基質と結合すると、ヘリカーゼドメインがアポ状態の伸びた状態(図3A)から基質を囲むような閉じた状態となることが明らかになりました(図3B)。具体的には non-canonical な RNA 結合ドメインである DUF283 ドメインが Hel1 を中心に150度回転し、PL ヘリックスがそれに伴って V 字型に変化します(図3B)。次に Dicer-2 はヘリカーゼドメインによる ATP の加水分解によって、基質を Platform-PAZ ドメインの RNA 結合ポケットに向かって移動させます。これをトランスロケーションと呼びます。トランスロケーション状態の構造では、基質は CRBD を介してさらに曲げられることにより、Platform-PAZ ドメインの RNA 結合ポケットに一直線に結合できることが明らかになりました(図3C)。基質がさらにトランスロケーションしてその末端が RNA 結合ポケットに結合すると、DP リンカーの一部の helix がほどけて伸びた状態となります(図3D)。DP リンカーは Dicer-2 のコア部分(Platform-PAZ‒RNase III)とヘリカーゼを繋ぐばねのような働きをします。この “ばね” が伸びることでコア部分がヘリカーゼに対して40度回転する構造変化を起こし、RNA 結合ポケットからちょうど21塩基長の位置が RNase III ドメインの活性部位と一致するようになります(図3D)。これらの立体構造から、Dicer-2 が正確に21塩基長で基質を切断するしくみが明らかになりました。
Dicer-2‒Loqs-PD は基質切断後、二本鎖 siRNA を放出し、二本鎖 siRNA は Dicer-2‒R2D2 と再結合することが知られています。基質切断後の Dicer-2‒Loqs-PD の立体構造では、DP リンカーの helix は伸びた状態から回復しており、CRBD は柔軟な構造をとるなど、二本鎖 siRNA の放出に適した構造となっていました(図3E)。次に二本鎖 siRNA が Dicer-2‒R2D2 と再結合すると、R2D2 と主に相互作用し、Dicer-2 の RNA 結合ポケットには結合していないことが明らかになりました(図3F)。また二本鎖 siRNA の熱力学的に安定な 5′ 末端はR2D2 のRBD2 とCTD に結合し、一方で熱力学的に不安定な 5′ 末端は溶媒に露出していました(図3F)。このように、Dicer-2‒R2D2 は二本鎖 siRNA を一方向で認識することで、Ago2 による不安定な 5′ 末端を含む siRNA の取り込みを促進していることが示唆されました。またヘリカーゼドメインにも二本鎖 siRNA が結合しており、基質を示していると考えられますが、基質の切断と二本鎖 siRNA の受け渡しのステップが一つの Dicer-2 で行われているのかは不明であり今後の研究が待たれます。
おわりに
Su らによる論文と筆者らによる論文は、クライオ電子顕微鏡によって、ショウジョウバエの siRNA 経路のスナップショットを捉えられることを示したものです。最近では、ショウジョウバエ Dicer-1‒LoqsPB (18) やヒト Dicer による miRNA 前駆体の切断機構 (19) が報告されており、Dicer の立体構造解析はますます盛んになることが期待されるでしょう。今回の研究を通して Dicer が複数のドメインを介して、反応をすすめる分子機構が明らかになったことは非常に面白く、このような研究に携われたことに深く感謝申し上げます。特に泊先生の2004年の論文と一致して、Dicer-2‒R2D2 が二本鎖 siRNA を認識しているのが立体構造から確認できたときと、自分が作った変異体の二本鎖 siRNA の末端認識のパターンが異なっていたときの感動は忘れられません。濡木理教授、西増弘志教授、泊幸秀教授をはじめとする先生方、そして携わってくださったすべての方々に感謝申し上げます。また、本稿の作製にあたって、平泉将浩助教、志甫谷渉助教、また濡木研究室の学生の方に多くのコメントを頂き、御礼を申し上げます。
文献
- Ghildiyal, M. & Zamore, P. D. Nat. Rev. Genet. 10, 94‒108 (2009).
- Liu, Q. et al. R2D2, Science 301, 1921‒1925 (2003).
- Jiang, F. et al. Genes and Development 19, 1674‒1679 (2005).
- Hartig, J. V. et al. EMBO J. 28, 2932‒2944 (2009).
- Tomari, Y. et al. Cell 116, 831‒841 (2004).
- Pham, J. W. et al. Cell 117, 83‒94 (2004).
- Miyoshi, K. et al. RNA 16, 506‒515 (2010).
- Tomari, Y. et al. Science 306, 1377‒1380 (2004).
- Schwarz, D. S. et al. Cell 115, 199‒208 (2003).
- Khvorova, A. et al. Cell 115, 505 (2003).
- Macrae, I. J. et al. Science 311, 195‒198 (2006).
- Liu, Z. et al. Cell 173, 1191–1203.e12 (2018).
- Wei, X. et al. Nat Plants 7, 1389‒1396 (2021).
- Wang, Q. et al. Science 374, 1152‒1157 (2021).
- Yamaguchi, S. et al. Nature 607, 393‒398 (2022).
- Su, S. et al. Nature 607, 399‒406 (2022).
- Hiraizumi, M., PSSJ Archives, 14, e099 (2021).
- Jouravleva, K. et al. Mol. Cell 82, 4049–4063.e6 (2022).
- Zapletal, D. et al. Mol. Cell 82, 4064–4079.e13 (2022).