概要
蛋白質のX線結晶構造解析において、蛋白質結晶の作製は必須のステップである。通常、百から千程度の結晶化条件をスクリーニングし、目的蛋白質が結晶化する条件を探索する。しかしながら、多くの蛋白質が結晶化スクリーニングを行った後でも結晶化せず、結晶構造決定のボトルネックとなっている。これまでに、結晶化しない蛋白質を結晶化させるための試みが数多く提案されてきた。例としては、相同な蛋白質のDNAシャッフリング(1)や対称的に多量体化させcrystal contact数を減少させる方法(2)などがある。それぞれの方法論には一長一短があり、どの方法が最良であるとは言えないが、本プロトコルでは比較的に広く用いられている、表面残基エントロピー減少法(Surface Entropy Reduction Method;SER法)について筆者の行った例と合わせて解説する。
イントロダクション
表面残基エントロピー減少法(SER法)は結晶化が困難な蛋白質試料を結晶化させる試みとして、バージニア大学のDerewendaらによって提案された(3)。現在、この方法は単純かつ強力であるため、広く用いられるようになってきている。これまでに20近い蛋白質の結晶化が報告されている(4)。蛋白質表面に存在する大きな親水性の残基は側鎖のコンフォメーションエントロピーが高いため、結晶化の際に比較的に大きなエントロピー障壁を越えなければならない。これらの残基をAlaなど小さな残基に変異させることによって、側鎖のコンフォメーションエントロピーを減少し、結晶化パッキングの際のエントロピー損失を減らす。また、GluやLysなど高いコンフォメーションエントロピーを持つ残基は同時に電荷を持つ残基でもある。このため、これらの残基を電荷的に中性な残基に変異させることは、結晶形成の際の好ましくない電荷相互作用を取り除く効果も期待できる。さらに、蛋白質表面の形状的に考えても、これらの変異によって平らな表面が出来るため、結晶化パッキングの際の分子間接触をより大きくできることが期待される。表面残基エントロピー減少法は、新規蛋白質の結晶化のみではなく、分解能の改善やモデル蛋白質として簡単に結晶化する系の構築を目指す場合などにおいても有用である。
Outer Surface Protein A(OspA)の結晶化変異体の構築(5)
OspAはボレリア菌由来のリポ蛋白質であり、ライム病を媒介するマダニへの感染に関与する。分子量31 kDa、273残基からなり、N末端のCysが脂質修飾されている。OspAの結晶構造は抗体の複合体としてこれまでに2つ報告された。この結晶構造から、2つのドメインが単層βシートによってリンクされたダンベル様構造であることが分かり、この単層βシートはβシートを研究する上で魅力的なモデルになり得ると我々は考えた。
しかしながら、OspA単独では結晶化せず、抗体との複合体で結晶化する必要がある。そこで我々はOspAの単層βシート領域をβシート研究のモデルシステムとして用いるために、OspAの結晶構造を簡便に決定できる方法の確立を目指した。これまでのNMR測定からN末端領域はフレキシブルであることが分かっていたため、N末端26残基を欠失させた変異体を構築し、結晶化スクリーングを行った。しかし、1700条件を超える結晶化スクリーニングを行ったにもかかわらず、結晶を得ることが出来なかった。OspAはLysとGlu残基が全251残基中62残基(25%)も含んでおり、これはDerewendaらがモデルケースとして用いたRhoGDIのLys、Glu含有率20%より多い。我々はOspA結晶化が困難な原因はこの異常に多い高エントロピー残基含有率にあると考えた。そこで、表面残基エントロピー減少法によって結晶化可能な変異体の構築を目指した。単層βシート領域をモデルシステムとして用いることが目的であるため、表面残基の変異はN末端もしくはC末端ドメインに導入した。
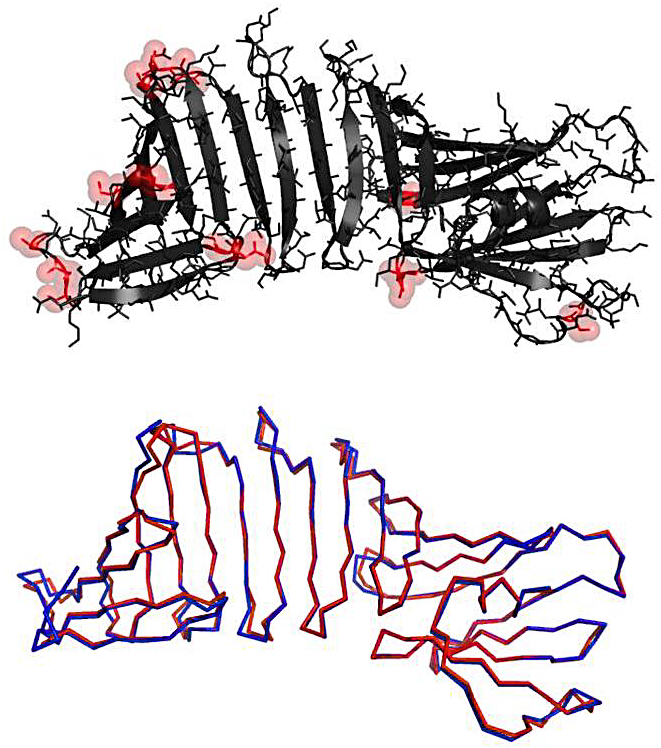
我々は最初に、これまでに報告されていた2種類の抗体複合体の結晶構造を詳細に観察した。その結果、2種類の結晶構造間では結晶パッキングが完全に異なっているにもかかわらず、3つの残基(Lys28, Glu196, Lys230)はどちらの結晶構造においても結晶中の分子間接触に関与していた。このことから、この三残基が結晶形成時に分子間接触に関与しやすい “Hot Spot” 残基であると考え、これらの残基をいくつかの組み合わせでアラニンに変異させた変異体を作製した。そのうち230位への変異は大腸菌発現において封入体形成が多くなったため、残りの2つの部位のAla変異体の結晶化スクリーニングを行った。しかし、野生型と同様にこの変異体も結晶の形成が観察されなかった。そこでさらに、OspA野生型の結晶構造(抗体との複合体)を観察し溶媒への露出が顕著な2残基をアラニンへ変異させた。この計4残基をアラニンに変異させた変異体も結晶化スクリーニングによって結晶を得ることが出来なかった。通常は、表面残基エントロピー減少法では1つの領域に対してアラニン変異を行い結晶化を目指すが、OspAでは高エントロピー残基であるLys、Gluが蛋白質表面の全領域に多く分布しているため、1つの領域に対する変異では不十分であることが考えられる。我々はさらに9残基への変異導入を検討した。初めの4残基はすでにアラニンに変異しているため、さらなるアラニン変異を導入することは溶解度の著しい減少が危惧された。そのため新たに加える9つの変異は極性残基のSerへ変異させた(図上に変位部位を示す)。この全部で13の残基を変異させた変異体は直ちに結晶化した。1536条件のハイスループット結晶化スクリーニング条件のうち、299条件で結晶化した(19.5%)。初期スクリーニング結晶化条件から良好な単結晶を得ることに成功し、この結晶から分解能1.15 Åの回折データを得ることが出来た。決定した変異体の結晶構造は野生型の結晶構造と驚くほど同一であった(RMSD:0.823 Å、図下)。変異を導入した13残基のうち、8残基が結晶構造中で分子間接触に関与していた。高分解能の結晶構造から、単層βシート領域の特徴的な水和構造が明らかとなり、この水和構造によって単層βシート構造の硬さがもたらされていることが示唆された。
著者たちはこの結晶化変異体をβシート研究のモデル蛋白質として用いて、これまでに多くのOspA変異体の結晶構造解析に成功してきている。
他の例として、筆者たちはデノボ蛋白質TOP7の構造解析における分解能の改善にSER法を用いた。TOP7は初めてデノボ設計されたα/β構造を持つ球状蛋白質であり、進化の配列への影響を受けていないため、モデル蛋白質として適している(6)。しかし、筆者たちが構造決定を試みたところ、分解能が低い結晶(3 Å以上)しか得られなかったためSER法を行い、追加の変異導入を経て、1.4 Åの構造決定に成功した。このようにSER法は非天然の蛋白質の構造決定にも有効である。
実験手順
1. 置換する残基
大きなコンフォメーション自由度がある、LysもしくはGlu。これらの残基は約90%の割合で蛋白質表面に位置する。これらの残基を低エントロピー残基である、Alaへ置換する。後述するが、Alaへの置換によって溶解度が減少するので、溶解度が問題となる場合は、Serなどの他の低エントロピー残基も検討する。
2. どこに変異を導入するか
ループやターン領域の高エントロピー残基を置換する。統計的にLys・Gluはαへリックスもしくはループ/ターン領域に多く存在するが、αへリックス領域の置換は主鎖が埋没しているため、あまり有効でない。相同性のある蛋白質の構造が既知である場合は、ホモロジーモデリングにより推定構造を構築し変異導入部位を決める(側鎖全体が溶媒へ露出している部位への変位が有効であると思われる)。構造が完全に未知の場合は、二次構造予測を用いる。通常は蛋白質配列中にLys・Gluが2つから3つ連続するパッチが数カ所存在し、その中の1つの部位をさせるが(例えばRhoGDIのGlu154/Glu155変異)、それでも結晶化に成功しない場合は複数のパッチに同時に変異を導入してみる。
3. 変異導入により懸念される問題点
A. 白質機能に重要な残基への変異導入によって機能が失われる可能性
PSI-BLASTなどにより保存性の高い残基を見つけることで、ある程度、機能に重要な残基を推定することが出来る。
B. 変異導入による溶解度の減少
低エントロピー残基としてまず選択肢としてAlaを考えるが、Alaは疎水残基であり、この変異によって溶解度の減少が懸念される。溶解度の減少が問題となる場合、Serなど他の親水的な低エントロピー残基を検討する。Derewendaらは他にもTyr、Thrへの置換を提案している(7)。
4. SERpサーバ
以上、手動による変異導入部位の検討方法を述べたが、最近この変異導入部位を自動で決定してくれるサーバが公開された(https://services.mbi.ucla.edu/SER/intro.php)(4)。
SERpサーバでは、まず問い合わせ配列の⑴二次構造予測(コイル領域が高スコア)、⑵連続した3残基のエントロピープロファイルを計算(高エントロピー残基が高スコア)、⑶PSI-BLASTによる保存残基の同定(保存されている残基は低スコア、Alaへの変異が見られる部位は高スコア)を行う。これらの計算結果を基に、最適な変異導入クラスター部位を提案してくれる。エントロピー障壁を越えるために、1つのクラスターパッチ中の変異はすべて同時に変異導入する必要がある。
筆者の行ったOspAへの表面残基変異がどの程度に予測可能かSERpサーバで計算をしたところ、サーバが提案した8つのクラスターのうち、意図的に除外した単層βシート領域とN-、C-末端以外の3クラスターが一致していた(変異した13残基のうちの5残基)(5)。このことから、このサーバは有効であると思われる。
工夫とコツ
変異導入法
一つの表面残基パッチに変異を導入する場合はQuikChange(Stratagene)などの単変異導入法を用いると簡便で良いが、複数の部位に同時に変異を導入する必要がある場合、複数の部位に同時に変異プライマーが結合できるKunkel法を用いると迅速に発現ベクターを構築できる。加えて、変異導入の副産物として、すべてに変異が導入されていないいろいろな組み合わせの変異体が得られるので、それら副産物も結晶化トライアルを行ってみる価値がある。
文献
- Keenan, R. et al., Proc. Natl. Acad. Sci. USA, 102, 8887–92 (2005)
- Banatao, R. et al., Proc. Natl. Acad. Sci. USA, 103, 16230–35 (2006)
- Derewenda, ZS., Structure, 12, 529–35 (2004)
- Goldschmidt, L. et al., Protein Sci., 8, 1569–76 (2007)
- Makabe, K. et al., Protein Sci., 15, 1907–14 (2006)
- Ito, Y. et al., Int. J. Mol. Sci., 23, 701 (2022)
- Cooper, DR. et al., Acta Crystallogr. D Biol. Crystallogr., 63, 636–45, (2007)
改訂履歴
2025年6月5日 改訂
- 著者所属の情報の更新
- 細かい文言の修正
- P.3にTOP7の構造解析の例に関する文章を追加
- P.4のSERpサーバのURLを更新
- 文献6)を追加し、元の文献6)を文献7)に変更